Marcin Kowiel, Dariusz Brzezinski, Miroslaw Gilski, Mariusz Jaskolski: Conformation-dependent restraints for polynucleotides: the sugar moiety, Nucleic Acids Research (48)(2) (2020) pp. 962-973
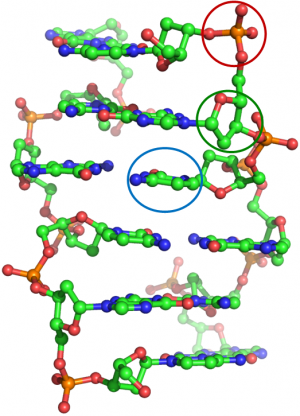
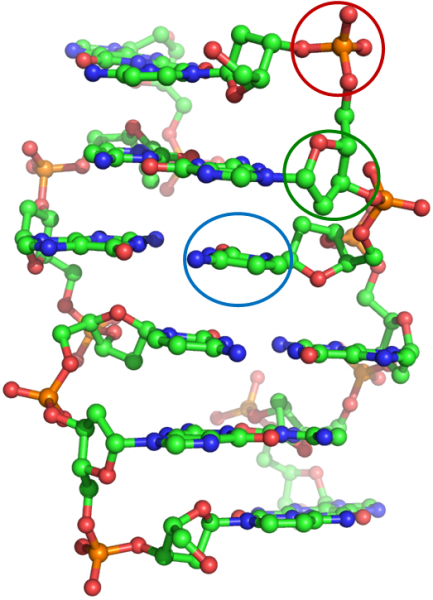
Budowę przestrzenną makromolekuł biologicznych, w tym kwasów nukleinowych, ustala się głównie na podstawie badania dyfrakcji rentgenowskiej ich kryształów. Polega to na wyznaczeniu położenia w przestrzeni każdego z wielu tysięcy atomów w ich strukturze. Część obliczeniowa tych badań zwykle boryka się z niewystarczającą liczbą danych doświadczalnych do udokładnienia tak gigantycznego modelu. Zaradzić temu może narzucenie na model więzów ograniczających swobodę jego parametrów geometrycznych, takich jak np. długości wiązań chemicznych. Takie biblioteki więzów musza być opracowane z najwyższą starannością, gdyż ewentualne błędy zostałyby „odziedziczone” przez kolejno udokładniane struktury. Nagrodzona praca jest częścią tryptyku, w którym opracowano zaawansowane (bo zależne od konformacji czyli typu struktury) więzy geometryczne dla kwasów nukleinowych. Więzy te wyznaczono na podstawie najdokładniejszych struktur krystalicznych komponentów kwasów nukleinowych zawartych w ogromnej bazie danych Cambridge Structural Database. Konkretnie, w omawianym artykule przedstawiono więzy dla części cukrowej (tj. rybozy), która jest najbardziej skomplikowanym fragmentem łańcuchów DNA czy RNA (oprócz rybozy w skład tych łańcuchów wchodzą jeszcze grupy fosforanowe oraz zasady nukleinowe). Na ilustracji przedstawiono fragment struktury DNA z zaznaczoną przykładowo grupą cukrową (kolor zielony), fosforanową (czerwony) oraz zasadą (niebieski). Nowa biblioteka więzów dostępna jest bez ograniczenia dla wszystkich zainteresowanych za pośrednictwem serwera RestraintLib.
Szczegóły: https://academic.oup.com/nar/article/48/2/962/5651323